
Introduction
돌연변이는 유전정보를 가지는 유전자의 염기서열이 바뀌게 되어 기존의 염기서열과 달라지는 현상을 의미합니다. 돌연변이의 결과물 중 하나가 우리가 잘 알고 있는 암(Cancer)입니다.
세포는 사멸 신호를 받으면 죽게 되는 세포사멸 시스템을 가지고 있습니다. 세포사멸 시스템은 생명체에 있어서 매우 중요한 시스템 중 하나입니다. 이 시스템에 문제가 생기게 되면 세포는 죽지 않고 계속 분열하여 끊임없이 증식하게 됩니다. 우리는 이러한 세포를 암이라고 부르고 있습니다.(정확하게는 한자리에서만 분열하는 세포를 종양이라고 하고, 여기저기 전이되어 마음대로 분열하는 세포를 악성종양 또는 암이라고 부르고 있습니다.) 이번에는 돌연변이 유발원과 돌연변이 종류, 돌연변이 복구 시스템, 그리고 암과 관련된 돌연변이에 대하여 알아보도록 하겠습니다.
돌연변이 유발원
유전정보를 가지고 있는 DNA의 염기서열에 변화를 주는 돌연변이 유발원은 크게 유도 돌연변이, 자연 돌연변이로 나눌 수 있습니다.
| 유도 돌연변이: 물리적 돌연변이, 유사염기 첨가, 삽입 물질, 화학적 돌연변이 유발원 |
◎ 물리적 돌연변이 유발원
- 자외선: DNA가 자외선에 노출되면 인접한 피리미딘 염기 사이에 공유결합이 형성하여 피리미딘 이량체를 형성할 수 있습니다. 이렇게 형성된 피리미딘 이량체는 DNA 복제 시, frmae-shift 돌연변이가 발생하여 DNA 염기서열이 바뀌게 됩니다. 피리미딘 이량체의 대표적인 예는 티민 이량체입니다.
*참고: DNA 염기에는 피리미딘 계열, 퓨린 계열이 있으며 피리미딘 계열은 티민(T), 시토신(C)이며 퓨린 계열은 아데닌(A), 구아닌(G)입니다.
- 방사선: 방사선의 높은 에너지 때문에 라디칼이 생성되어 DNA를 박살 냅니다.(DNA내의 공유결합을 파괴합니다.)
- 열: DNA 이중가닥을 형성하는 수소결합은 열에 의해 파괴되어 DNA가 벌어지게 됩니다. 이때, 물분자가 DNA 사이로 침투하게 되고, 침투한 물분자가 DNA의 염기에 변형을 유도할 수 있습니다.
- 전이인자: 사람의 DNA의 45%를 차지하는 전이인자가 염색체의 다른 위치로 이동할 때, 중요한 유전자를 파괴할 수 있습니다. 전이인자는 enhencer, poly A tail 신호 제공, microRNA 서열 제공, 프로모터 등의 다양한 기능을 수행하고 있고, 전이인자가 제공한 새로운 프로모터에 의해 새로운 유전자 발현 산물이 발생하기 때문에 생물진화의 수단으로 생각되고 있습니다.
◎ 유사 염기 첨가: DNA의 염기와 유사한 염기를 세포에 처리할 경우 DNA 사이에 유사 염기가 끼어들어가게 되고, 끼어들어간 유사 염기가 엉뚱한 염기들과 상보 가닥을 형성하게 되고, 결국 염기 치환 돌연변이가 발생할 수 있습니다. 대표적인 유사 염기는 BrdU(5-Bromodeoxyuracil), 2-Aminopurine이 있습니다.
◎ 삽입 물질: 이중나선인 DNA의 내부에는 염기쌍들이 수소결합을 이루면서 차곡차곡 쌓여 있습니다. 쌓여있는 염기쌍들 사이에 소수성 결합으로 끼어들 수 있는 납작한 분자들을 삽입 물질이라고 하며, 삽입 물질들에 의해 frme-shift 돌연변이가 발생할 수 있습니다. 삽입물 질의 대표적인 예로는 EtBr이 있습니다.
*참고: 삽입 물질인 EtBr은 자외선에 오렌지색 형광을 나타내기 때문에 주로 DNA를 염색하는 데에 사용됩니다. 젤 전기영동 후 EtBr을 첨가하면 DNA 밴드 사이로 삽입이 되고, 자외선을 쬐어주면 DNA 밴드를 확인할 수 있습니다.
◎ 화학적 돌연변이 유발원: 염기에 변화를 일으켜 염기 치환 돌연변이를 일으키는 화학적 돌연변이 유발원을 의미합니다. 하이드록 시화, 탈아미노, 알킬화, 탈퓨린 반응 등등이 포함됩니다.
- Hydroxyl amine: 시토신(Cytosine)을 N-4-hydroxyl cytosine으로 바꾸어주어, C와 G 염기쌍을 T와 A염기쌍으로 바뀌게 하는 결과를 가져옵니다.
- HNO2: HNO2(아질산)은 탈이미노 반응을 유발하여 시토신(Cytosine)을 U(우라실)로 바꾸어 놓습니다. 그 결과 C와 G 염기쌍을 T와 A의 염기쌍으로 바뀌는 염기 치환 돌연변이를 일으키게 합니다.
- Alkylating agent: DNA의 염기에 alkyl(알킬) 기를 붙여주는 역할을 하고, 결국 염기 치환 돌연변이를 일으키게 합니다. 대표적으로 EMS(ethyl methane sulfonate), ENU(Ethyl nitrosourea), MMS(methyl methane sulfonate) 등이 있습니다.
돌연변이 종류
돌연변이의 종류는 크게 coding 지역의 돌연변이와 non-coding 지역의 돌연변이, 염색체 구조 돌연변이, 염색체 수 이상 돌연변이로 나눌 수 있습니다.
◎ coding region 돌연변이
- 첨가 또는 결실 돌연변이(frame-shift 돌연변이)

정상 DNA에 염기가 첨가 또는 결실로 인하여 염기서열이 바뀌게 되고 위와 같이 정상적인 단백질이 형성이 되지 않는 돌연변이를 frame-shift 돌연변이라고 합니다. frmae-shift 돌연변이로 인하여 정상 단백질에 비하여 길이가 짧아질 수도 있고 또는 길어질 수도 있습니다.
- 치환(하나의 염기가 다른 염기로 치환되는 돌연변이)
치환에는 치환되는 염기의 종류에 따라 크게 2가지로 나뉩니다.
- 전위(transition, 염기 치환): Purine(퓨린) 계열 염기가 다른 Purine(퓨린) 계열 염기로 또는 Pyrimidine(피리미딘) 계열의 염기가 다른 Pyrimidine(피리미딘) 계열의 염기로 치환되는 경우로 예를 들어, A(아데닌)이 G(구아닌)으로, T(티민)이 C(시토신)으로 치환되는 경우를 전위라고 합니다.
- 전좌(transversion, 염기 치환): Purine(퓨린) 계열 염기가 Pyrimidine(피리미딘) 계열의 염기로 또는 Pyrimidine(피리미딘) 계열의 염기가 Purine(퓨린) 계열의 염기로 치환되는 경우로 예를 들어, A(아데닌)이 C(시토신)으로, A(아데닌)이 T(티민)으로 치환되는 경우를 전좌라고 합니다.
치환으로 인해 일어나는 돌연변이는 침묵 돌연변이, 중립 돌연변이, 미스 센스 돌연변이, 난센스 돌연변이로 정리를 할 수 있습니다.
- 침묵 돌연변이(Silent mutation): 변화된 염기서열이 변화 전과 동일한 아미노산을 암호화하는 코드일 경우를 의미하는 돌연변이로, 생명체에는 영향을 주지 않습니다.
- 중립 돌연변이(Neutral mutation): 염기서열의 변화에 의해 번역된 아미노산이 바뀌었지만, 단백질의 기능에는 크게 영향을 주지 않는 경우의 돌연변이로, 대부분 비슷한 성질의 아미노산으로 바뀐 경우입니다.
- 미스 센스 돌연변이(Missense mutation): 치환된 염기서열로 인하여 다른 성질의 아미노산으로 바뀌어 정상적인 단백질의 기능을 못하게 하는 돌연변이를 의미합니다.
- 넌센스 돌연변이(Nonsense mutation): 변화된 염기서열이 stop codon이 되는 경우로, 단백질의 합성이 초반에 끝나버려 기능이 상실된 단백질을 만들어버리는 돌연변이를 의미합니다.

◎ non-coding region 돌연변이
- 전사 인자 결합 부위, 프로모터 등의 전사 조절 부위에 일어나는 돌연변이를 의미하며, 유전자의 전사 자체를 막을 수 있는 돌연변이입니다.
◎ 염색체 구조 돌연변이
- 결실(deletion): 염색체의 일부가 사라진 경우를 의미합니다.
- 역위(inversion): 염색체의 일부가 뒤집혀서 들어간 경우로, 원래의 배열 순서가 달라지게 되는 경우를 의미합니다.
- 중복(duplication): 동일한 염색체의 일부가 중복되어 늘어난 절편이 삽입된 경우를 의미합니다.
- 전좌(translocation): 염색체의 일부가 원래 있어야 할 자리가 아닌 다른 자리로 이동된 경우를 의미합니다.
◎ 염색체 수 이상 돌연변이
- 배수성 돌연변이(Polylioidy): 완전한 반수체의 염색체 세트를 더 가지게 되는 경우로, 전체 염색체의 수는 제대로 복사되어서 증폭되었지만 유사 분열이 제대로 일어나지 않아 염색체 세트를 더 가지게 되는 돌연변이를 의미합니다. (2n이 아니라, 3n 또는 4n 등의 염색체 세트를 가지게 된 경우) 대표적인 예로 씨 없는 수박이 있습니다.

- 이수성 돌연변이(Aneuploidy): 감수분열 중 염색체의 비분리 형상에 의해 염색체의 수가 2n보다 적거나 많게 되는 경우를 의미합니다.
돌연변이 복구 시스템
생명체의 돌연변이 복구 시스템은 크게 손상 복귀, 손상 제거, 손상 무시 시스템으로 3가지가 있습니다.
◎ 손상 복귀(DNA reversal)
- 광재 활성화(Photoreactivation): 자외선으로 생성된 티민 이량체는 photolyase 단백질이 인식하여 가시광선 영역의 빛(blue light)을 이용하여 티민 이량체 결합을 파괴하여 정상적인 DNA로 복구합니다.
- 메틸기 제거: Alkyltransferase의 효소는 구아닌의 5번 위치에 결합한 메틸기 또는 알킬기를 직접 제거를 할 수 있어, 원래의 염기로 복구해 줍니다.
- Ligation: DNA에 phosphodiester 결합이 끊어져 nick이 발생된 경우, ligase 효소가 nick을 제거하여 원래의 DNA로 복구합니다.
◎ 손상 제거(DNA removal)
- Base exicision repair: 탈아미노 반응에 의하여 생성된 비정상적인 염기를 DNA glycosylase가 인식하고 염기와 당 사이의 결합을 절단하여 AP(apyrimidine) 자리를 만듭니다. AP자리를 AP endonuclease가 인식하여 AP자리의 phosphodiester 결합을 끊습니다. 그리고 DNA polymerase의 5'->3' exonuclease 활성을 통하여 손상된 지역을 제거하고, 정상 DNA로 합성합니다. 끝으로 발생된 nick은 DNA ligase에 의해 제거되며 DNA의 복구가 완료됩니다.
*참고: Base exicision repair에 사용된 DNA polymerase는 DNA polymerase I 입니다.
- Mismatch repair: MutS는 새로 합성된 DNA에서 잘못된 염기쌍이 있는지 확인하고, 잘못된 염기쌍이 있을 경우, MutL과 함께 메틸화 된 5'-GATC-3'가닥을 찾고, MutH를 활성화시켜 메틸화 되지 않은 5'-GATC-3'쪽의 가닥을 절단합니다. 그 뒤, MutD helicase가 결합하여 절단된 가닥을 푸는 과정을 가집니다. 풀어진 가닥은 exonuclease가 절단 지점에서부터 시작하여 오류가 있는 가닥을 분해한 다음 DNA polymerase에 의해 다시 DNA가 합성되고 DNA ligase에 의해 발생된 nick을 제거하여 마무리합니다.
*참고: Mismatch repair에 사용되는 DNA polymerase는 DNA polymerase III 입니다.
| DNA는 일정시간이 지난후에 유전자 발현 조절 및 자신이 만든 제한 효소에 의해 DNA가 절단되는 것을 막기위해 메틸화를 합니다. 주형 DNA는 이미 메틸화가 되어있는 상태이고 새로 합성된 DNA는 일정시간이 지나기전에는 메틸화가 안되어 있기때문에 메틸화의 유무로 주형가닥과 새롭게 합성된 가닥을 구분합니다. |
- Nucleotide excision repair: 티민 이량체에 의해 DNA의 왜곡이 심할 경우 사용되는 돌연변이 복구 시스템입니다. UvrAB가 DNA를 따라 움직이면서 돌연변이가 일어난 부위를 찾아냅니다. 돌연변이 부위를 감지하면 UvrA는 떨어지고, UvrC 단백질이 결합하여 돌연변이가 일어난 부위에 3' 방향으로 뉴클레오티드를 3~4개를 분해하고, 5'방향으로 8개가량의 뉴클레오티드를 분해하여 gap을 형성시키는데, UvrC가 gap을 형성하는 동안 UvrD helicase가 DNA를 풀어주는 역할을 해줍니다. UvrABC에 의해 생긴 gap은 DNA polymerase가 채워주고 생긴 nick은 DNA ligase에 의해 제거되며 DNA 복구는 완료됩니다.
*참고: Nucleotide excision repair에 사용된 DNA polymerase는 DNA polymerase I 이며, UvrABC를 exinuclease라고 합니다.
◎ 손상 무시(Damage tolerance)
DNA의 손상이 심각할 경우 사용되는 복구 시스템으로, Recombinational repair, SOS repair가 있습니다.
- Recombinational repair: DNA polymerase3가 DNA를 복제 시, DNA에 티민 이량체와 같은 손상된 부위를 만나면 약 800 염기 가량을 뛰어넘어 gap을 형성시키고 다시 복제를 이어 나갑니다. RecA 단백질은 반대편의 주형가닥을 들고 와서 gap을 메꾸어 줍니다. RecA에 의해 생성된 반대편의 gap은 DNA polymerase1, DNA ligase에 의해 메꾸어집니다. 이런 기작을 통해 복제된 DNA는 문제가 없게 되지만 여전히 주형가닥에는 티민 이량체와 같은 손상된 부위가 남아있게 되는데 이는 추후에 다른 복구 기작으로 수선됩니다.
- SOS repair (Error-prone repair): 대장균에 과도한 돌연변이가 발생하여 도저히 DNA 복제를 할 수 없을 때 진행되는 복구 기작으로, DNA 수선에 연관된 유전자들의 operator 서열인 SOS box에 결합된 LexA 단백질을 RecA가 활성화되어 분해를 촉진시킵니다. LexA 단백질이 분해됨에 따라 SOS repair 유전자들의 발현이 촉진됩니다. LexA 단백질의 분해로 인해 세포분열을 차단하는 sulA, sulB 유전자가 발현되어 세포분열을 차단하며, Nucletide excision repair에 관여하는 Uvr 유전자가 발현되기 시작합니다. 약 40분이 지나도 복구가 되지 않으면 translesion DNA 합성경로가 활성화되어 DNA polymerase4(DinB), DNA polymerase5(UmuCD)에 의해 손상된 부위에 무작위로 염기를 끼어넣어서 DNA 합성을 진행합니다. 이 결과로 DNA주형과 전혀 다른 DNA의 합성이 일어나게 됩니다.
*참고: SOS repairs는 DNA에 너무 많은 손상이 있으며, 수선이 불가능할 경우 발생되는 것으로 대장균이 DNA 손상에 대해 어쩔 수 없는 상황일 때 발생되는 시스템입니다.
세포사멸(Apoptosis)과 암과의 관계
세포는 외부에서 세포사멸 신호(Fas ligand) 또는 DNA가 너무 망가졌을 경우 세포를 죽게 하는 시스템을 가지고 있으며 우리는 이 시스템을 세포사멸(Apoptosis)라고 하고 있습니다. 세포사멸은 생명의 발생과정 중 매우 중요한 역할을 하고 있는데 예를 들어, 사람이 발생하면서 손의 세포 일부를 죽여 손가락을 형성, 올챙이가 성장하면서 꼬리를 없애는 경우에 사용되는 시스템입니다.
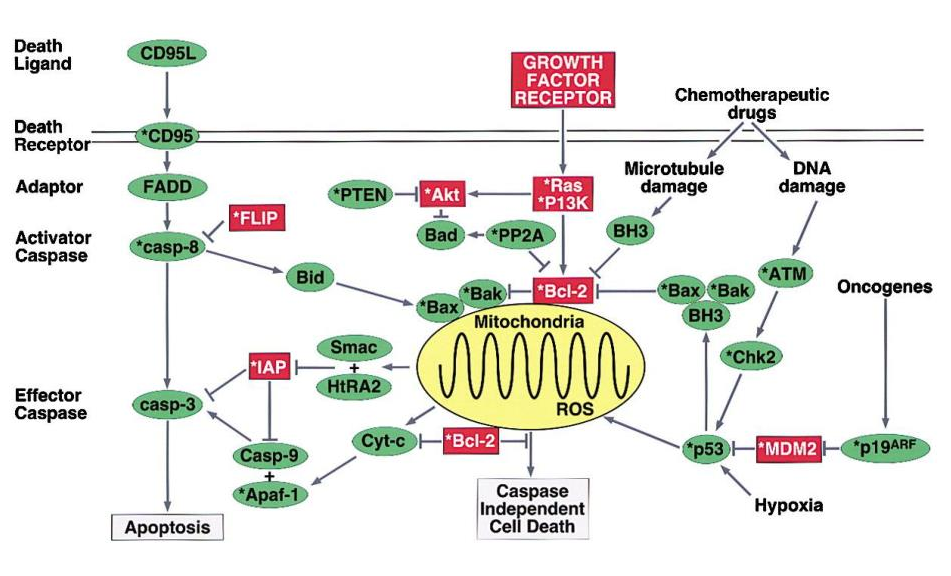
세포사멸은 Fas ligand(CD95L)로 부터 또는 DNA damage에 의해 P53 단백질의 활성화로부터 시작하여 일련의 과정을 거쳐 일어나게 됩니다.
돌연변이로 인하여, Fas 수용체, Bid이 발현되지 않거나, 미토콘드리아에 있는 Bcl-2가 기존보다 많이 있는 경우에는 Fas ligand(세포사멸 신호)가 있어도 세포사멸이 일어나지 않아 암세포로 될 가능성이 높아집니다. 그리고 암 억제 단백질로 유명한 P53역 시 돌연변이로 인하여 발현되지 않거나 또는 제대로 된 기능을 못하게 될 경우 역시 암세포로 될 가능성이 매우 높아집니다.
세포성장 및 분열과 암과의 관계
세포사멸에 관한 돌연변이 말고도 세포성장 및 분열에 관한 돌연변이로 인하여 암이 발생되기도 하는데, 정상 유전자이지만 돌연변이로 인하여 발암유전자(oncogene)로 변하는 proto-oncogene, 염색체 구조 돌연변이가 여기에 속합니다.
◎ Proto-oncogene의 돌연변이
- PDGF(platelet-derived growth factor) 관련 돌연변이: 활성화된 혈소판에서 생성되고 방출되며, 혈관의 형성과 성장에 관여하는 성장인자입니다. Simian sarcoma virus의 DNA에는 PDGF B의 유전자인 c-Sis의 염기서열이 매우 유사한 v-Sis 유전자를 가지고 있습니다. Simian sarcoma virus에 감염되면, virus의 v-Sis의 과량 발현으로 인하여 표적 세포에 성장 신호를 지속적으로 전달하게 되어 암을 유발합니다.(v-Sis를 oncogene이라고 합니다.)
- EGFR(epidermal growth factor receptor) 관련 돌연변이

EGFR은 성장 인자가 결합하기 전에는 수용체 도메인이 tyrosine kinase 도메인을 저해하고 있다가 성장 인자가 결합하게 되면 구조의 변화를 통해 tyrosine kinase의 활성이 나타나게 되고(인산화됩니다.) 세포 분열, 세포 사멸 억제 등의 활동이 시작되게 됩니다. avian erythroblastosis virus(AEV)에 감염되면 AEV가 가지고 있는 v-erbB1 유전자는 수용체 도메인의 결실로 인하여 tyrosine kinase 도메인이 항상 활성을 지니고 있어 세포에 감염이 되면 암을 일으키게 합니다.
- HER2(human epidermal growth factor receptor) 관련 돌연변이: 돌연변이로 인해 erB2 유전자의 과량 발현으로 HER2 단백질들이 많이 만들어져 세포질 내로 과도한 세포 분열 신호를 전달하게 되어 암을 일으키게 합니다.
◎ 염색체 구조 돌연변이
- 중복: 유전자의 중복으로 인하여 특정 단백질(성장인자 수용체, 세포사멸 억제 관련 단백질 등등)이 과량으로 발현되어 암이 발생합니다.
- 결실: 조절 유전자의 결실, 특정 유전자의 조절 도메인의 결실 등으로 인하여 세포분열이 계속되어 암이 발생합니다.
- 역위: 새로운 위치로 이동한 조절 유전자가 position effect로 인하여 제대로 발현되지 못해 암이 발생합니다. 예를 들어 조절 유전자가 염색체의 centromere로 이동되었을 경우 제대로 발현되지 못하게 되는 경우를 의미합니다.
- 전좌: 상호 전좌를 통해 enhencer 서열이 이동되어 특정 유전자의 과량 발현을 일으키거나, 서로 다른 위치에 있던 유전자들이 상호 전좌의 결과로 같이 연결되어 융합 단백질을 형성하여 세포 분열이 계속되는 경우를 의미합니다.
마무리
돌연변이로 인하여 염기서열이 하나라도 바뀌게 되었을 경우, 특히 이 부분이 유전정보를 가지고 있는 엑손(exon)이라면 엄청난 악영향을 가져올 수 있습니다. 그리고 이 돌연변이가 세포사멸, 세포 성장 및 억제에 관련된 유전자에 일어나게 되면 암이 발생되게 됩니다. 항암치료를 통해서 암을 치료를 할 수 있을지도 모르지만, 암에 걸리기 전에 돌연변이 유발원에 최대한 노출되지 않아야 하고, virus에 의해서도 암에 걸릴 수도 있으니 virus도 조심해야 한다고 생각합니다. 실은 매일 우리 몸에서는 몇 개의 암이 생기고 DNA 복구 시스템에 의해서 암이 사라지는 과정이 반복된다고 합니다. 최대한 돌연변이 유발원에 노출되지 않아 암을 예방하도록 합시다. 이상입니다.
'Biology' 카테고리의 다른 글
| 생명공학의 발전과 환경에 미치는 영향 (0) | 2020.07.23 |
|---|---|
| 맥주: 인간이 이용한 최초의 생명공학 과정의 부산물 (0) | 2020.07.21 |
| 혈액형과 유전의 상관관계 (2) | 2020.07.17 |
| DNA를 증폭시키는 방법과 응용: PCR(중합효소연쇄반응), DNA fingerprinting (2) | 2020.07.16 |
| DNA에서 다양한 유전자 발현이 생기는 이유: Alternative splicing (0) | 2020.07.16 |








최근댓글